Gli anticorpi rappresentano attualmente la classe principale di terapeutici proteici, con oltre 160 prodotti autorizzati a livello globale e un valore di mercato previsto che raggiungerà i 445 miliardi di dollari nei prossimi cinque anni.
Il processo di sviluppo degli anticorpi si articola generalmente in due fasi principali: la scoperta degli anticorpi che si legano a un epitopo specifico e la successiva maturazione dell’affinità e ottimizzazione clinica di questi anticorpi.
Attualmente, l’identificazione di anticorpi specifici per epitopi si basa su metodi di immunizzazione animale o screening di librerie di anticorpi per identificare molecole candidate in grado di legarsi a un target desiderato, seguiti da una mappatura degli epitopi. Questi metodi sono laboriosi, richiedono molto tempo e possono fallire nell’individuare anticorpi che interagiscono con l’epitopo terapeuticamente rilevante.
Gli sforzi nella progettazione computazionale degli anticorpi si sono generalmente concentrati sulla seconda fase di ottimizzazione del processo di sviluppo, come il campionamento di loop CDR alternativi per migliorare le affinità o l’uso del design di sequenze Rosetta per migliorare le regioni interattive.
Recentemente, sono stati addestrati reti di deep learning basate su struttura e sequenza per progettare varianti di sequenza di anticorpi nuovi, ma questi metodi necessitano di un anticorpo inizialmente legante dal quale trarre ispirazione per l’ottimizzazione.
Nonostante i rapidi progressi nella progettazione di proteine leganti, esistono poche o nessuna metodologia computazionale in grado di eseguire la prima fase della progettazione di anticorpi, rendendo così risolvibile il problema della progettazione de novo (senza omologia a un anticorpo esistente) degli anticorpi.
Un approccio ideale per progettare anticorpi de novo dovrebbe abilitare:
1. Targeting di qualsiasi epitopo specificato su un target di interesse.
2. Focalizzazione del campionamento sui loop CDR, mantenendo la sequenza e la struttura del framework vicine a un framework terapeutico altamente ottimizzato.
3. Campionamento di posizionamenti rigidi alternativi dell’anticorpo progettato rispetto all’epitopo.
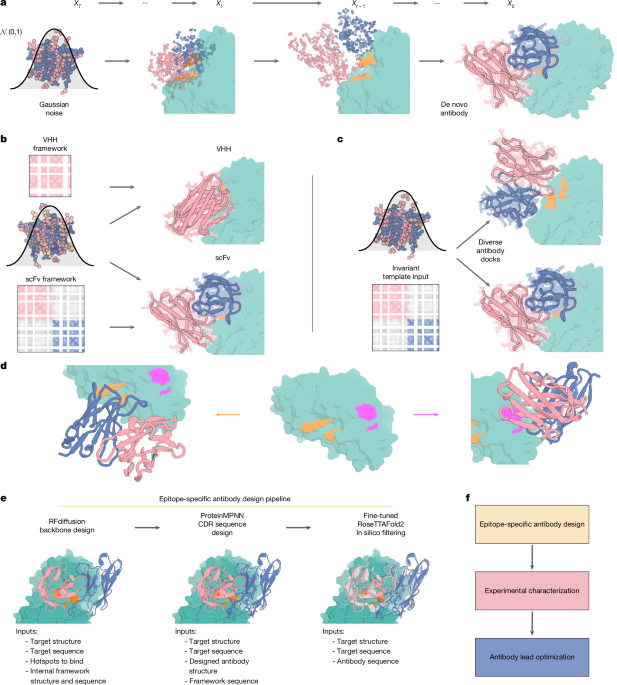
Abbiamo ipotizzato che una versione specializzata di RFdiffusion, perfezionata su strutture anticorpali, dovrebbe essere in grado di progettare interfacce mediate da CDR de novo, data la diversità e qualità delle interfacce de novo che RFdiffusion può progettare. La nostra proposta si basa sul fatto che la termodinamica sottostante alla formazione dell’interfaccia è la medesima.
RFdiffusion utilizza la rappresentazione del frame RF2 dell’AlphaFold2 dei backbones proteici comprendenti le coordinate Cα e l’orientamento N-Cα-C per ciascun residuo. Durante l’addestramento, si utilizza un piano di corruzione che, nel corso di un numero prestabilito di “passaggi temporali”, corrompe i frame proteici verso distribuzioni precedenti casuali. Le coordinate Cα vengono corrotte con rumore gaussiano tridimensionale e le orientazioni dei residui con movimento browniano su SO3.
Durante l’addestramento, viene campionata una struttura del Protein Data Bank (PDB) e viene applicato un passaggio di corruzione casuale. RFdiffusion predice la struttura de-noised e minimizza un errore quadratico medio fra la struttura vera e la predizione.
Al momento dell’inferenza, viene campionata una distribuzione di residui casuale, e RFdiffusion de-noises iterativamente per generare nuove strutture proteiche. Abbiamo perfezionato RFdiffusion prevalentemente su strutture di complessi anticorpali.
Ad ogni passo di addestramento, la struttura dell’anticorpo viene corrotta per consentire la specificazione della sequenza e della struttura del framework in tempo reale.
Poiché è desiderabile che la posizione rigida (dock) tra anticorpo e target venga progettata da RFdiffusion insieme alle conformazioni dei loop CDR, la struttura del framework è fornita in un modo invariato rispetto al frame globale durante l’addestramento. Utilizziamo la ‘template track’ di RF2/RFdiffusion per fornire la struttura del framework come matrice bidimensionale di distanze e angoli diedrali tra ogni coppia di residui, una rappresentazione dalla quale possono essere ricapitolate con precisione strutture tridimensionali.
Il framework e i template del target non codificano le loro posizioni relative nello spazio tridimensionale. In questo lavoro, abbiamo mantenuto fissa la sequenza e la struttura della regione di framework, concentrandoci sulla progettazione dei CDR e sul posizionamento rigido generale dell’anticorpo verso il target. Abbiamo addestrato RFdiffusion con una caratteristica “hotspot” codificata one-hot, che fornisce una frazione di residui con cui i CDR dell’anticorpo interagiscono, affinché all’inferenza possiamo indirizzare gli anticorpi verso un sito specifico, che ci si riferisce come “epitopi”.
Con questo regime di addestramento, RFdiffusion è in grado di progettare strutture anticorpali che corrispondono da vicino alla struttura della struttura di framework di input e mirano all’epitopo specificato con loop CDR nuovi.
Dopo il passaggio di RFdiffusion, utilizziamo ProteinMPNN per progettare le sequenze dei loop CDR. Gli anticorpi progettati fanno interazioni diverse con l’epitopo target e differiscono significativamente da sequenze nel dataset di addestramento. Non c’è correlazione tra la similarità del dataset di addestramento e il successo del legame.
I pipeline di progettazione generano tipicamente una vasta gamma di soluzioni per qualsiasi sfida di design. Un modo efficace per filtrare le proteine progettate e le interfacce che hanno maggiori probabilità di avere successo sperimentalmente è fondato sulla somiglianza della struttura progettata con la struttura prevista da AlphaFold2 per la sequenza progettata, a ciò che spesso viene fatto riferimento come ‘autoconsistenza’. Tuttavia, nel caso degli anticorpi, AlphaFold2 non riesce a prevedere con precisione le strutture anticorpali, impedendo l’uso come filtro in un pipeline di progettazione anticorporale.
Abbiamo cercato di migliorare il filtraggio della progettazione perfezionando RoseTTAFold2 su strutture anticorpali. Per semplificare la previsione della struttura anticorpale, abbiamo fornito informazioni durante l’addestramento sulla struttura del target e sulla posizione dell’epitopo a cui si lega l’anticorpo.
Questo approccio ha rivelato che il fine-tuning di RF2 è in grado di distinguere robustamente le vere coppie anticorpo-antigene dalle coppie di decoy e predice spesso con precisione le strutture dei complessi anticorpo-antigene.
Quando questa rete RF2 fine-tuned viene utilizzata per ripredire la struttura degli VHH progettati da RFdiffusion, una frazione significativa viene predetta come legante in modo quasi identico alla loro struttura progettata. Inoltre, le analisi in silico di cross-reattività hanno dimostrato che gli VHH progettati da RFdiffusion vengono raramente previsti come leganti a proteine non correlate.
Gli VHH che sono previsti con fiducia di legarsi al loro target progettato sono previsti di formare interfacce di alta qualità.
Ci siamo inizialmente concentrati sulla progettazione di anticorpi monodimensionali (VHH) prodotti da camelidi. Ad oggi, due terapie basate su VHH sono state approvate dalla FDA con numerosi trial clinici in corso. Anche se i VHH hanno meno loop CDR (tre rispetto ai sei delle proteine anticorpali convenzionali), l’area media di interazione di un VHH è molto simile a quella di un anticorpo, suggerendo che un metodo capace di progettare VHH potrebbe essere altrettanto adeguato per la progettazione di anticorpi.
Abbiamo scelto un framework VHH umanizzato ampiamente utilizzato come base delle nostre campagne di progettazione, progettando VHH per una serie di target pertinenti a malattie come C. difficile TcdB, influenza H1 emaglutinina, virus respiratorio sinciziale e SARS-CoV-2 dominio di legame (RBD) e IL-7Rα.
I design computazionali filtrati sono stati esaminati ad alta attraverso di visualizzazione su superficie di lievito oppure a bassa attraverso con espressione di Escherichia coli seguita da risonanza plasmonica di superficie (SPR).
I più alti leganti a affinabilità per il sito III del virus respiratorio sinciziale, emaglutinina di influenza, RBD e TcdB sono stati misurati con costanti di dissociazione (Kd) variabili quindi mostrati nei risultati.
Per TcdB, l’epitopo target è l’interfaccia di Frizzled, per la quale non esistono anticorpi o VHH indirizzati a questo sito nel PDB.
Il migliore VHH progettato ha mostrato una costante di dissociazione di 5.5 μM per il RBD e 260 nM per il TcdB. Tutti i dati hanno dimostrato che l’abilità di RFdiffusion per progettare VHH in grado di interagire specificamente con l’epitopo target.
Inoltre, abbiamo utilizzato la criomicroscopia elettronica (cryo-EM) per caratterizzare strutture di VHH progettati contro l’emaglutinina di influenza, dimostrando che essa si lega in modo efficace al bersaglio complesso.
Una campagna di affinamento che ha utilizzato un sistema di replicazione a DNA con tasso di errore ortogonale ha permesso una rapida maturazione dell’affinità degli anticorpi originari.
Alla fine, abbiamo identificato con successo scFv completi che legano il complesso PHOX2B-HLA-C*07:02, stabilendo collegamenti specifici.
Questi risultati sono significativi perché dimostrano che la progettazione de novo di anticorpi affiancata a metodologie sperimentali possono rivoluzionare la scoperta e lo sviluppo di anticorpi nel campo della terapia medica.
Oggi, progettiamo anticorpi che possono rispondere a epitope specifici, le cui applicazioni cliniche sono già promettenti e aprono la porta a future terapie targeted.
Fonte dell’immagine:nature